
DNA Lab
"Quando finalmente interpretati, i messaggi genetici codificati all'interno delle nostre molecole di DNA forniranno le risposte finali alle basi chimiche dell'esistenza umana" James D. Watson.
La piattaforma tecnologica DNA LAB del CEINGE fornisce molteplici strumenti per "leggere" questi messaggi genetici e per "scriverne" di nuovi grazie ai suoi due settori Servizio di Sequenziamento di Acidi Nucleici di Sanger e Servizio di Sintesi di Oligonucleotidi.
SERVIZIO DI SEQUENZIAMENTO DI SANGER
Il servizio di sequenziamento Sanger del CEINGE, grazie alla sua lunga esperienza nei processi automatizzati di sequenziamento con il metodo di Sanger, offre la massima flessibilità anche per campioni che richiedono soluzioni su misura, in tubi ed in piastre.
Garantiamo l'interpretazione di sequenze ripetute, siano esse brevi ripetizioni in tandem (STR o microsatelliti) o ripetizioni a intervalli brevi e lunghi (SINE e LINE), nonché riarrangiamenti strutturali.
La qualità analitica garantita per la singola lettura è precisione del 99% per le prime 700 basi per sequenziamento singolo e un errore ogni 10000 basi per progetti di sequenziamento.
Il nostro scopo principale è fornire ai nostri utenti, indipendentemente dalle obiettive difficoltà presentate dallo specifico progetto di campionamento o sequenziamento, il nostro supporto per dare loro il miglior risultato possibile.
I NOSTRI SERVIZI DI SEQUENZIO DEL DNA
Servizio di Sequenziamento Premix Tube: campioni di DNA risospesi in acqua, plasmidi e PCR, premiscelati con primers specifici ed organizzati in provette Eppendorf da 1,5 ml (in numero massimo di otto) o in strisce da otto tubi.
Il templato deve essere costituito da DNA purificato in una delle seguenti concentrazioni:
- DNA plasmidico fino a 10 kb: 100 ng / μl, volume finale di 8 μl PIU’ 8 μl del Primer specifico ad una concentrazione 2 μM. Assicurarsi che il volume totale della miscela DNA / Primer sia di almeno 16 μl. Il campione di DNA deve essere quantizzato al NanoDrop.
- DNA plasmidico oltre 10 kb: 100 ng / μl, volume finale di 16 μl PIU’ 8 μl del Primer specifico ad una concentrazione 2 μM. Assicurarsi che il volume totale della miscela di DNA / Primer sia di 24 μl. Il campione di DNA deve essere quantizzato al NanoDrop.

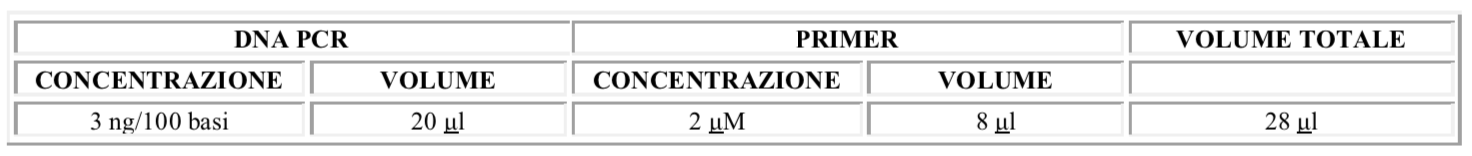
- Prodotti di PCR purificati: 3 ng ogni 100 bp in un volume finale di 20 μl PIU’ 8 μl del Primer specifico ad una concentrazione 2 μM. Assicurarsi che il volume totale della miscela di DNA / Primer sia di 28 μl. Il campione di DNA deve essere quantizzato al NanoDrop.
- OGNI ORDINE DEVE ESSERE ACCOMPAGNATO DA UN MODELLO MOD SS_01 DEBITAMENTE COMPILATO O DA STAMPA DELLA RICHIESTA A SCOPI ESPLICATIVI.
- Contrassegnare ogni provetta con il numero del campione corrispondente a quello inserito nel modulo MOD SS_01 (da richiedere al personale della facility) o con numero della richiesta, usando un pennarello indelebile.
- Contrassegnare la prima provetta della striscia di otto provette usando un pennarello indelebile e organizzare i campioni nello stesso modo indicato nel modulo MOD SS_02 (da richiedere al personale della facility).
- Nel caso in cui i campioni siano > 30kbp e / o contengano tratti difficili da leggere, si prega di riportarlo nel modello MOD SS_01. Speciali condizioni di PCR verranno adottate allo scopo di ottenere i migliori risultati di sequenziamento possibili.
- Far recapitare i campioni da sequenziare a temperatura ambiente nei nostri laboratori.
- I risultati verranno inviati via e-mail entro il secondo giorno successivo a quello di ricevimento dell’ordine.
- Una ripetizione gratuita è prevista in caso di guasto tecnico
- Servizio di sequenziamento Tube: fornisce sequenze di campioni di DNA risospesi in acqua, plasmidi e PCR, usando primers standard del servizio ed organizzati in provette Eppendorf da 1,5 ml (in numero massimo di otto) o in strisce da otto tubi.
- Il templato deve essere costituito da DNA ad una delle seguenti concentrazioni:
- DNA plasmidico fino a 15 kb: 100 ng / μl, volume finale di 20 μl.

-
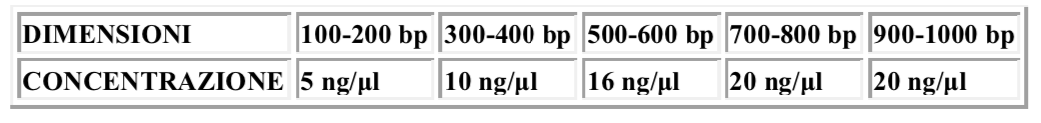
PCR prodotti non purificati: 100-200 bp: 5 ng / μl; 300-400 bp: 10 ng / μl; 500-600 bp: 16 ng / μl; > 1000bp 20ng / μl volume finale di 20 μl.
-
Prodotti di PCR purificati: 3 ng ogni 100 bp in un volume finale di 20. Il campione di DNA deve essere quantizzato al NanoDrop

- OGNI ORDINE DEVE ESSERE ACCOMPAGNATO DA UN MODELLO MOD SS_01 DEBITAMENTE COMPILATO O DA STAMPA DELLA RICHIESTA A SCOPI ESPLICATIVI.
-
Contrassegnare ogni provetta con il numero di campione corrispondente inserito nel modulo MOD SS_01 (da richiedere al personale della facility) o con numero della richiesta, usando un pennarello indelebile.
-
Contrassegnare la prima provetta della striscia di otto provette usando un pennarello indelebile e organizzare i campioni nello stesso modo indicato nel modulo MOD SS_02 (da richiedere al personale della facility).
-
Quantizzare i templati da purificare mediante gel di agarosio o QIAXEL utilizzando comunque un idoneo marker DI PESO MOLECOLARE E DI CONCENTRAZIONE. L’immagine prodotta dal QIAxcel, di contro, deve contenere un massimo di tredici campioni, così da permetterne chiara interpretazione e deve riportare la data inserita di default dalla strumentazione.
-
Nel caso in cui i campioni siano > 30kbp e / o contengano tratti difficili da leggere, si prega di riportarlo nel modello MOD SS_01. Speciali condizioni di PCR verranno adottate allo scopo di ottenere i migliori risultati di sequenziamento possibili.
-
Far recapitare i campioni da sequenziare a temperatura ambiente nei nostri laboratori.
-
I risultati verranno inviati via e-mail entro il secondo giorno successivo a quello di ricevimento dell’ordine.
-
Una ripetizione gratuita è prevista in caso di guasto tecnico.
-
DNA stampo e primers ordinati vengono conservati per due settimane.
-
La purificazione della PCR è inclusa nel prezzo.
SERVIZIO DI SEQUENZIAMENTO IN PIASTRA
Servizio di Sequenziamento Premix Plate: campioni di DNA risospesi in acqua, plasmidi e PCR, premiscelati con primers specifici ed organizzati in piastra.
Il templato deve essere costituito da DNA purificato in una delle seguenti concentrazioni:
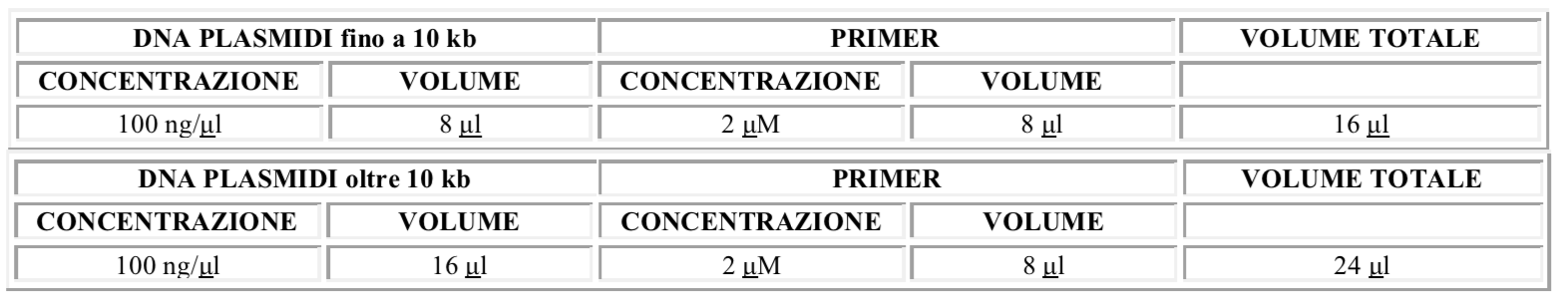
- DNA plasmidico fino a 10 kb: 100 ng / μl, volume finale di 8 μl PIU’ 8 μl del Primer specifico ad una concentrazione di 2 μM. Assicurasi che il volume totale della miscela DNA / primer sia di almeno 16 μl. Il campione di DNA deve essere quantizzato al NanoDrop.
- DNA plasmidico oltre 10 kb: 100 ng / μl, volume finale di 16 μl PIU’ 8 μl del Primer specifico con una concentrazione di 2 μM. Assicurasi che il volume totale della miscela DNA / primer sia di 24 μl. Il campione di DNA deve essere quantizzato al NanoDrop.
-
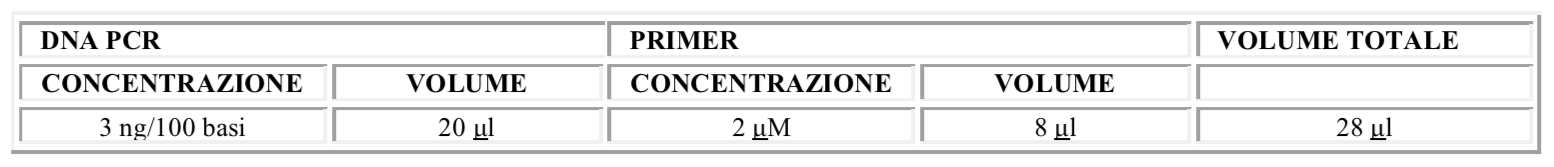
Prodotti di PCR purificati: 3 ng ogni 100 bp di volume finale di 20 μl PIU’ 8 μl del tuo Primer personalizzato con una concentrazione di 2 μM. Assicurarsi che il volume totale della miscela di DNA / primer sia di 28 μl. Il campione di DNA deve essere quantizzato al NanoDrop.
-
OGNI ORDINE DEVE ESSERE ACCOMPAGNATO DA UN MODELLO MOD SS_01 DEBITAMENTE COMPILATO O DA STAMPA DELLA RICHIESTA A SCOPI ESPLICATIVI.
-
Si prega di organizzare i campioni nella piastra nello stesso modo indicato nel modulo MOD SS_02 (da richiedere al personale della facility).
-
Nel caso in cui i campioni siano > 30kbp e / o contengano tratti difficili da leggere, si prega di riportarlo nel modello MOD SS_01. Speciali condizioni di PCR verranno adottate allo scopo di ottenere i migliori risultati di sequenziamento possibili.
-
I risultati verranno inviati via e-mail entro il secondo giorno successivo a quello di ricevimento dell’ordine.
-
Una ripetizione gratuita è prevista in caso di guasto tecnico.
-
La concentrazione deve essere misurata mediante NanoDrop e normalizzata attraverso la piastra.
-
Le piastre possono contenere diversi tipi di campioni di DNA disposti in colonne dalla prima alla dodicesima e dall'alto verso il basso.
-
La dimensioni dei prodotti di PCR non devono variare di più di un fattore 3.
-
La posizione H12 della piastra deve essere mantenuta libera per il controllo di qualità interno.
-
Sigillare le piastre con strisce a 8 cappucci per prevenire la perdita di materiale.
-
Far recapitare i campioni da sequenziare a temperatura ambiente nei nostri laboratori.
-
Servizio di Sequenziamento Plate: fornisce sequenze di campioni di DNA risospesi in acqua usando primers standard del servizio. Campioni in piastra.
Il templato deve essere costituito da DNA in una delle seguenti concentrazioni:
-
DNA plasmidico fino a 15 kb: 100 ng / μl, volume finale di 20 μl.

-
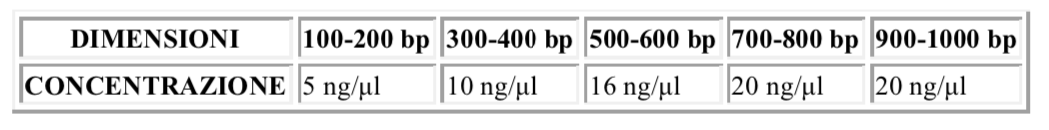
PRODOTTI non purificati: 100-200 bp: 5 ng / μl; 300-400 bp: 10 ng / μl; 500-600 bp: 16 ng / μl;> 1000bp 20ng / μl volume finale di 20 μl.
-
Prodotti di PCR purificati: 3 ng ogni 100 bp in un volume finale di 20. Il campione di DNA deve essere quantizzato al NanoDrop.

-
OGNI ORDINE DEVE ESSERE ACCOMPAGNATO DA UN MODELLO MOD SS_01 DEBITAMENTE COMPILATO O DA STAMPA DELLA RICHIESTA A SCOPI ESPLICATIVI.
-
Si prega di organizzare i campioni nella piastra nello stesso modo indicato nel modulo MOD SS_02 (da richiedere al personale della facility).
-
Nel caso in cui i campioni siano > 30kbp e / o contengano tratti difficili da leggere, si prega di riportarlo nel modello MOD SS_01. Speciali condizioni di PCR verranno adottate allo scopo di ottenere i migliori risultati di sequenziamento possibili.
-
I risultati verranno inviati via e-mail entro il secondo giorno successivo a quello di ricevimento dell’ordine.
-
Una ripetizione gratuita è prevista in caso di guasto tecnico.
-
DNA stampo e primers ordinati vengono conservati per due settimane.
-
Quantizzare i templati mediante gel di agarosio o QIAXEL utilizzando comunque un idoneo marker DI PESO MOLECOLARE E DI CONCENTRAZIONE. L’immagine prodotta dal QIAxcel, di contro, deve contenere un massimo di tredici campioni, così da permetterne chiara interpretazione e deve riportare la data inserita di default dalla strumentazione.
-
La concentrazione deve essere normalizzata attraverso la piastra.
-
Le piastre possono contenere diversi tipi di campioni di DNA disposti in colonne dalla prima alla dodicesima e dall'alto verso il basso.
-
La dimensioni dei prodotti di PCR non devono variare di più di un fattore 3.
-
La posizione H12 della piastra deve essere mantenuta libera per il controllo di qualità interno.
-
I prodotti PCR devono essere inviati liquidi in un volume totale di 28 μl.
-
Sigillare le piastre con strisce a 8 cappucci per prevenire la perdita di materiale.
-
Far recapitare i campioni da sequenziare a temperatura ambiente nei nostri laboratori.
-
Quantizzare i templati mediante gel di agarosio o QIAXEL o mediante lettura spettrofotometrica così da assicurare risultati accurati.
-
Walking Primer Service, Sequenza a singolo o doppio filamento mediante walking primer.
PRIMERS STANDARD
E’ possibile utilizzare i primers più comuni per i vettori più frequenti. L’elenco completo è indicato nella tabella in basso.
SEQUENZE CON PRIMERS CUSTOM
E’ possibile utilizzare per effettuare la reazione di sequenza primers propri, seguendo le indicazioni seguenti:
- la concentrazione del primer deve essere 2 µM;
- la quantità del primer deve essere di 30 µl per la prima reazione e 4 µl per ogni reazione ulteriore;
- la lunghezza del primer deve essere compresa tra i 18 ed i 25 nucleotidi;
- il contenuto in “GC” del primer deve essere uguale o maggiore al 50%;
- la temperatura di melting del primer deve essere compresa tra i 50 °C ed i 60 °C;
- condizioni che si discostino di molto da quanto indicato vanno riportate nel modello MOD SS_01.
SERVIZIO SINTESI OLIGONUCLEOTIDI
Gli oligonucleotidi sono molecole di DNA o RNA corte a filamento singolo. Hanno una vasta gamma di applicazioni in biochimica, biologia, diagnostica molecolare, genomica e altri esperimenti di biologia molecolare. Gli oligo possono essere anche modificati grazie al loro legame chimico con una varietà di molecole quali gruppi fosfato alle estremità 5' o 3' per consentirne il loro inserimento in un plasmide o la loro estensione; Fluorofori e / o Quencher per il loro uso come sonde; gruppi tiolo, amino ed altri gruppi reattivi che consentano un legame covalente di molecole funzionali come gli enzimi o con altri linker e spaziatori di diversa funzionalità.
La piattaforma tecnologica SERVIZIO SINTESI OLIGONUCLEOTIDI del CEINGE opera da più di vent'anni nella progettazione e produzione di oligonucleotidi, garantendo sempre ai suoi utenti prodotti di elevata qualità e purezza.
Tutte le molecole prodotte sono infatti controllate senza costi aggiuntivi mediante MALDI-TOF, siano esse di medie dimensioni o più lunghe o che abbiano modifiche intramolecolari ed esterne.
Gli oligonucleotidi vengono forniti grezzi o desalati mediante precipitazione in etanolo. A richiesta dell'utente e per requisiti tecnici specifici, l'oligo viene fornito dopo purificazione mediante HPLC, cartucce a fase inversa (RP-HPLC) o PAGE (purificazione mediante elettroforesi su gel di polyacrylammide).
La piattaforma di sintesi di oligonucleotide del CEINGE fornisce i seguenti servizi:
Sintesi di DNA grezzo e desalato Scale di sintesi 40 nmol-200 nmol-1 μm
• Sintesi di DNA non modificato
• Sintesi di DNA modificato
• Sonde di DNA marcati
• Altre modifiche concordabili
Sintesi di RNA grezzo e desalato Scale di sintesi 40 nmol-200 nmol-1 μm
• Sintesi di RNA non modificato
• Sintesi di RNA modificato
• Sintesi di oligonucleotidi di RNA lunghi
• Sonde di RNA marcati
• Altre modifiche concordabili
Sintesi di oligonucleotidi scala di sintesi 10 μm, grezzi e desalati
Purificazione mediante HPLC (lunghezza massima di oligonucleotidi 40 basi)
Purificazione mediante PAGE
